Differential expression and core genes mining of mitophagy-related genes in stomach adenocarcinoma
-
摘要:目的
探讨线粒体自噬相关基因(MRGs)在胃腺癌(STAD)中的差异表达并挖掘核心基因。
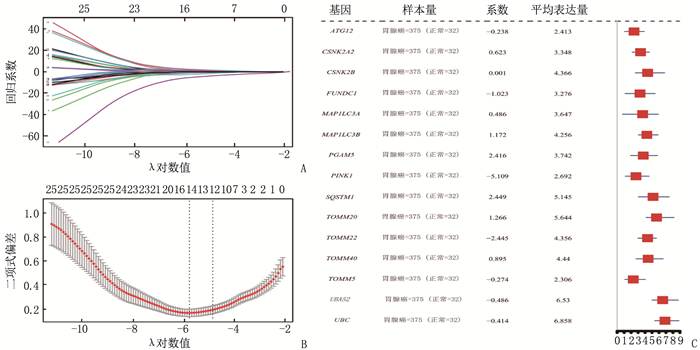
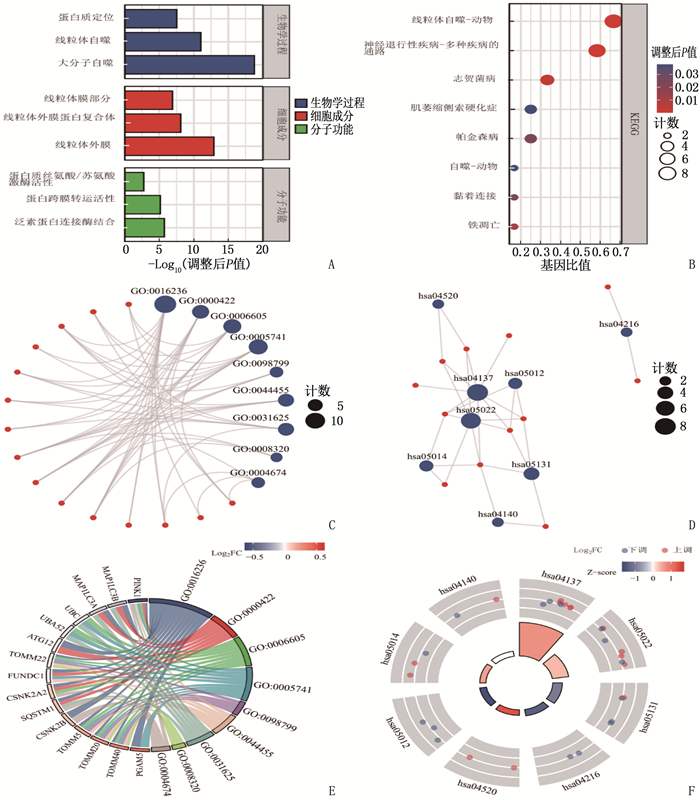
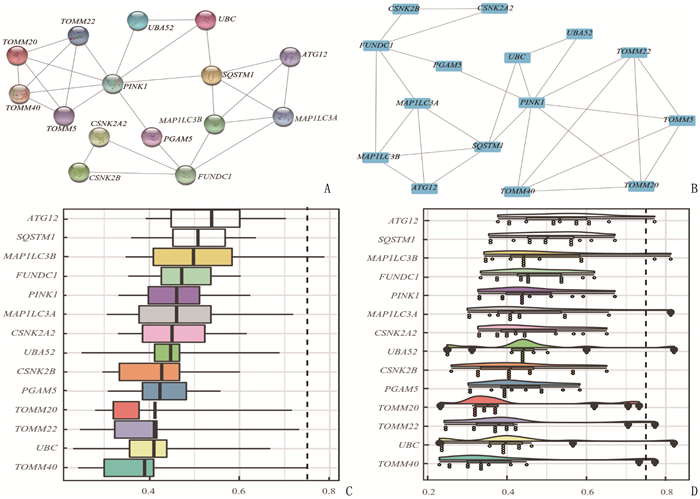
方法从基因表达综合数据库(GEO)和癌症基因组图谱(TCGA)数据库获得STAD患者的临床样本信息, 基于基因集富集分析(GSEA)网站和基因卡片(GeneCards)数据库获取有统计学意义的MRGs共26个; 基于最小绝对值收缩与选择算子(LASSO)回归构建MRGs在STAD中的预后模型,筛选出线粒体自噬预后相关基因(MPRGs)。通过基因本体论(GO)和京都基因与基因组百科全书(KEGG)富集分析,获得关键基因参与的生物学过程和通路; 利用STRING数据库构建MPRGs的蛋白质-蛋白质相互作用(PPI)网络,并应用Cytoscape软件进行可视化。
结果基于LASSO回归构建的预后模型中共筛选出15个MPRGs, 即ATG12、CSNK2A2、CSNK2B、FUNDC1、MAP1LC3A、MAP1LC3B、PGAM5、PINK1、SQSTM1、TOMM20、TOMM22、TOMM40、TOMM5、UBA52、UBC, 均为STAD的危险因素; 15个MPRGs中, UBC、UBA52基因对STAD的进展和预后影响更大, PGAM5表达与STAD的发生显著相关, ATG12基因与其他基因的功能相似性得分最高; PPI网络分析结果显示, PINK1蛋白与其他蛋白的相互作用最多。
结论15个MPRGs在STAD的发生与发展中起重要作用,或可作为STAD基因检测、治疗的靶点和STAD预后的独立预测工具。
Abstract:ObjectiveTo explore differential expression of mitophagy-related genes (MRGs) in stomach adenocarcinoma(STAD) and to investigate related core genes mining.
MethodsGene Expression Omnibus (GEO) and The Cancer Genome Atlas (TCGA) databases to obtain clinical sample information of patients with STAD. A total of 26 MRGs with statistical significance were obtained based on the Gene Set Enrichment Analysis (GSEA) website and GeneCards database. A prognostic model of MRGs in STAD was constructed based on Least Absolute Shrinkage and Selection Operator (LASSO) regression, and mitochondrial autophagy prognostic genes (MPRGs) were screened out. Through gene Ontology (GO) and Kyoto Encyclopedia of Genes and Genomes (KEGG) enrichment analysis, key genes involved in biological processes and pathways were obtained. Protein-protein interaction(PPI) network was established by means of STRING database, and the Cytoscape software was utilized for visualization.
ResultsFifteen MRGs were selected in the LASSO regression model: ATG12, CSNK2A2, CSNK2B, FUNDC1, MAP1LC3A, MAP1LC3B, PGAM5, PINK1, SQSTM1, TOMM20, TOMM22, TOMM40, TOMM5, UBA52 and UBC, which were risk factors for STAD. Among 15 MPRGs, UBC and UBA52 genes had more impact on progression and prognosis of STAD, and the expression of PGAM5 was significantly correlated with the occurrence of STAD. ATG12 gene had the highest functional similarity score with other genes. PPI network analysis showed that PINK1 protein had the most interactions with other proteins.
ConclusionA total of 15 MPRGs play important roles in the occurrence and development of STAD, and may be used as targets for STAD gene detection, treatment and independent prognostic tools for STAD.
-
乳腺癌侵袭性强、转移率较高、预后较差, 一般方法难以治疗。常用的治疗方法包括手术、化疗、放射治疗、内分泌治疗和分子靶向治疗等,所以寻找抗肿瘤药物的靶点和生物标志物仍然至关重要[1]。α1, 6-岩藻糖基转移酶由岩藻糖基转移酶8(FUT8)基因编码, FUT8可以将岩藻糖基转移到N-糖链上天冬酰胺连接的N-乙酰氨基葡萄糖残基的第6位,参与糖蛋白中N-连接的低聚糖的生物合成[2]。FUT8也与疾病密切相关, FUT8与乳腺癌的不良预后密切关联,具体原因仍有待探究[3]。
1. 材料与方法
1.1 TIMER平台和Oncomine平台分析FUT8在各类肿瘤中的表达情况
肿瘤免疫分析数据库(TIMER, https://cistrome.shinyapps.io/timer; TIMER2.0, http://timer.comp-genomics.org和Oncomine( https://www.oncomine.org
1.2 Kaplan-Meier Plotter平台分析生存情况
Kaplan-Meier Plotter( http://kmplot. com/analysis/
1.3 TIMER平台分析免疫细胞浸润情况
依托TIMER的Gene模块分析FUT8与乳腺癌各个分型中B细胞、CD4+T细胞、CD8+T细胞、巨噬细胞和DC细胞浸润的关系,并根据Cor值评价其相关性; 依托TIMER2.0平台的Immune Gene模块分析FUT8与Treg细胞和NK细胞浸润的关系,并根据Rho值来评价相关性。Cor或Rho值的绝对值> 0且P < 0.05被视为有相关性且差异有统计学意义。
1.4 FUT8与其他因子的相关性分析
利用TIMER2.0和bc-GenExMiner( http://bcgenex.ico.unicancer.fr/
1.5 免疫组织化学验证
取内蒙古医科大学附属医院2018—2020年就诊的经病理确诊的乳腺癌和腺病(良性对照)石蜡切片共40例进行FUT8的免疫组织化学染色,FUT8兔单克隆抗体购于英国Abcam公司(货号ab191571, 工作浓度1∶500); EDTA修复液(货号MVS-0099)、动物非免疫血清(货号SP KIT-B3)、即用型免疫组化试剂盒(货号KIT-9921)、DAB显色试剂盒(货号DAB-0031)均购于福建迈新公司。实验步骤按照各试剂说明书操作,并以扁桃体组织染色作为阳性对照, PBS替代一抗作为阴性对照。癌细胞和乳腺细胞胞质棕黄色染色且染色细胞数 > 10%被判定为阳性。
2. 结果
2.1 FUT8在乳腺癌以及其他癌症中的表达情况
FUT8的免疫组织化学染色结果显示, FUT8大多数定位于胞质, FUT8在20例乳腺癌组织中17例为阳性,表达率为85%, 在20例腺病组织中均为阳性,表达率为100%, 见图 1。
TIMER平台和Oncomine平台显示,与正常组织相比,FUT8在乳腺癌以及多种类型癌组织在转录组水平上均有上调,差异有统计学意义(P < 0.01), 说明FUT8可能在乳腺癌中发挥一定的作用,有望成为乳腺癌的标志物之一,见图 2。
另外,在Oncomine平台以“Breast Cancer”和“Triple Negative Status”为关键词进行筛选,探讨三阴乳腺癌和非三阴乳腺癌中的FUT8表达。Farmer数据库中FUT8表达的分组依据Basal样和Luminal样乳腺癌, TCGA数据库中的表达的分组依据为是否为ERBB2/ER/PR阴性状态。FUT8的表达在三阴和非三阴乳腺癌间的差异有统计学意义(P < 0.05), 见图 3。
2.2 FUT8对于生存预后的影响
在乳腺癌的研究中,通常将总生存时间(OS)和无复发生存时间(RFS)作为评价治疗效果的指标。本研究在Kaplan-Meier Plotter平台中发现, FUT8水平的变化与乳腺癌的预后效果存在相关性,乳腺癌患者中, FUT8呈低表达的患者OS和RFS较差,说明乳腺癌患者中FUT8的低表达可能对患者的生存以及肿瘤转移有不利影响,见图 4。
同时,本研究对乳腺癌的各类临床特征进行分组并进行OS和RFS的分析,分别统计分期、淋巴结转移、TP53突变情况、ER状态、PR状态、HER2状态以及乳腺癌内在亚型中不同条件下FUT8水平的变化对于生存和预后的影响。结果显示, FUT8的高表达会根据分期、淋巴结转移、TP53状态、ER水平、HER2水平改善乳腺癌患者的生存和预后(HR < 1)。同时, TP53突变型和PR阳性患者中, FUT8的高表达是患者生存和预后的危险因素(HR > 1)。在luminal A亚型以及HER2亚型的患者中, FUT8水平对于预测预后具有较高的价值,见表 1。
表 1 FUT8在乳腺癌不同临床特征中的生存预后情况临床特征 总生存时间 无复发生存时间 n HR P n HR P 分期 1期 175 0.61(0.24~1.58) 0.300 397 1.33(0.80~2.20) 0.270 2期 443 0.65(0.42~0.99) 0.043 1 177 0.57(0.45~0.72) < 0.001 3期 586 0.71(0.53~0.96) 0.026 1 300 0.86(0.70~1.06) 0.150 淋巴结转移 阳性 452 0.56(0.40~0.80) 0.001 1 656 0.72(0.60~0.87) < 0.001 阴性 726 0.60(0.42~0.84) 0.003 2 368 0.81(0.69~0.95) 0.011 TP53突变 野生型 197 0.49(0.26~0.93) 0.026 273 0.75(0.49~1.170 0.210 突变型 130 0.57(0.27~1.21) 0.140 188 1.72(1.07~2.76) 0.024 ER状态 阳性 754 0.75(0.54~1.05) 0.098 2 633 1.15(0.98~1.34) 0.080 阴性 520 0.62(0.44~0.88) 0.007 1 190 0.78(0.32~0.98) 0.029 PR状态 阳性 156 2.15(1.02~4.53) 0.039 926 1.30(0.98~1.73) 0.072 阴性 148 0.75(0.46~1.22) 0.240 925 0.74(0.57~0.96) 0.022 HER2状态 阳性 420 0.67(0.46~0.97) 0.032 882 0.88(0.69~1.11) 0.270 阴性 1459 0.56(0.45~0.69) < 0.001 4 047 0.67(0.59~0.75) < 0.001 内在亚型 Basal亚型 404 0.69(0.45~1.06) 0.090 846 1.13(0.90~1.42) 0.290 luminal A亚型 794 0.58(0.42~0.81) 0.001 2 277 0.80(0.67~0.96) 0.014 luminal B亚型 177 1.16(0.80~1.67) 0.440 1 491 0.86(0.71~1.03) 0.100 HER2+亚型 111 0.50(0.27~0.91) 0.021 315 1.35(0.94~1.93) 0.110 2.3 FUT8与乳腺癌的免疫细胞浸润水平相关性
采用TIMER和TIMER2.0平台的免疫细胞浸润分析模块,在乳腺癌的3个内在亚型(basal亚型、HER2+亚型和luminal亚型)分别探讨了FUT8表达与各类免疫细胞(B细胞、CD4+T细胞、CD8+T细胞、巨噬细胞、DC细胞、Treg细胞、NK细胞)浸润水平的关系,见图 5。在basal亚型中, FUT8的水平与CD8+T细胞、Treg细胞、NK细胞和巨噬细胞的浸润呈正相关; 在HER2+亚型中, FUT8的水平与CD4+T细胞和Treg细胞的浸润呈正相关; 在luminal亚型中,除Treg细胞外, FUT8表达水平与各类免疫细胞浸润无显著相关性(P > 0.05)。
由于巨噬细胞在肿瘤进展过程中的特殊作用,本研究额外在TIMER2.0平台上分析了不同亚型乳腺癌中FUT8与各个分型巨噬细胞浸润之间的相关性,见图 6。在basal、HER2、luminal A 3个亚型中, FUT8的表达水平与M2型巨噬细胞的浸润呈正相关。
2.4 FUT8与免疫检查点以及免疫细胞标志物的相关性
本研究通过TIMER2.0和bc-GenExMiner平台分析了FUT8与一些常见的免疫检查点以及免疫细胞(B细胞、Treg细胞、DC细胞、MDSC细胞、巨噬细胞)相关标志物之间的相关性,见图 7。在乳腺癌basal亚型和HER2亚型中, FUT8的表达与大多数标志物的表达呈正相关(P < 0.05), 在luminal亚型中呈负相关(P < 0.05)或无显著相关性(P > 0.05)。
3. 讨论
乳腺癌是女性最常见肿瘤,发病率高,治疗棘手,且预后不理想。手术、化疗、放射治疗、内分泌治疗等均为乳腺癌的主要治疗方法,但是对于缺少ER、PR和HER2表达的三阴乳腺癌患者,现有治疗方法不能减缓肿瘤进展,而且也不利于提高患者生活质量。乳腺癌的诊断和治疗策略逐渐成为近年来的研究热点,肿瘤分子标志物检测以及基于标志物的免疫疗法逐渐兴起,对于乳腺癌的诊断和治疗至关重要。
N-连接糖基化是内质网和高尔基体中的对新合成蛋白的修饰过程。N-连接的糖基化过程是由寡糖转移酶在内质网中启动的,在进入内质网腔的新合成的新生蛋白中,寡糖转移酶将多糖醇中的一个14糖核心多糖转移到N-X-T/S基序的天冬酰胺残基(其中N是天冬酰胺, X是除脯氨酸之外的任何氨基酸, S是丝氨酸,T是苏氨酸)。然后,在糖基化蛋白被转移到细胞膜前,其在内质网和高尔基体中修剪并进一步处理核心多糖[6]。一旦糖基化失调,蛋白质就被转运到胞浆,并迅速被降解。例如,在恶性肝病的发生和发展过程中, FUT8的活性显著升高,从而导致某些血清糖蛋白的α-1, 6-岩藻糖含量升高[7]。本研究通过生物信息学工具发现,乳腺癌和正常组织中,FUT8的表达差异有统计学意义(P < 0.05), 表明FUT8具有作为乳腺癌发生和发展的诊断基因的潜力。
研究[3]发现, FUT8可以在TGF-β诱导的上皮间质转化(EMT)过程中上调,从而增强肿瘤的侵袭和迁移能力。肿瘤的侵袭能力在一定程度上可以预示患者的预后情况,而本研究通过生物信息学工具却发现, FUT8水平的上调在大多数临床癌症类型中都有利于患者生存和预后,只对TP53的突变和PR阳性的患者不利,由于TP53与肿瘤细胞的增殖有着密切关系,推测FUT8和TP53的突变存在某种联系,有待于实验进一步验证。
肿瘤细胞和肿瘤免疫微环境(TME)之间的双向交流对正常组织稳态和肿瘤生长都是至关重要的,与肿瘤发生、进展和患者预后密切相关[8]。免疫细胞是肿瘤免疫微环境的组分之一,其中肿瘤相关巨噬细胞(TAMs)是肿瘤发生和进展的重要调节因素。大量研究[9]表明, TAMs在肿瘤进展的多个方面发挥着重要作用。本研究探讨了在乳腺癌中FUT8的水平与免疫细胞的浸润关系,结果发现M2型巨噬细胞的浸润与FUT8表达水平相关,同时M2型巨噬细胞的标志物CD163的表达也与FUT8呈正相关,说明FUT8在乳腺癌中可能参与了由M2型巨噬细胞主导的免疫抑制过程。同时,本研究也发现,在乳腺癌Treg细胞的浸润也与FUT8的表达水平相关。Treg细胞对不同环境刺激的反应具有复杂调节作用,研究[10-11]表明,不同类型的肿瘤中Treg细胞的增加对患者总生存率的影响也是不同的,与上文中FUT8对于生存和预后的影响结果也有一定的吻合,具体机制有待于进一步研究。
本研究还发现,在basal亚型中,各类免疫检查点的表达水平和FUT8呈正相关,但是TIGIT的配体PVR(又称CD155)水平与FUT8呈负相关, PVR在肿瘤组织中过度表达,与肿瘤的侵袭和迁移有密切联系[12]。NK细胞是先天免疫反应的淋巴细胞,其特征是在破坏肿瘤细胞中发挥作用,其激活受到多种因素调控[13]。研究[14]表明, PVR和NK细胞表面的TIGIT结合后会抑制NK细胞的功能。basal亚型中FUT8与NK细胞浸润和NK细胞标志物水平的正相关关系也验证了这一点。
本研究通过生物信息学工具研究了FUT8在乳腺癌以及其他肿瘤中的表达情况,以及其与乳腺癌免疫细胞浸润的关系和在不同临床特征下患者的预后情况,最后利用免疫组织化学实验进行初步验证。可见乳腺癌中有FUT8的表达; 乳腺癌中FUT8水平的升高和降低可能预示着更好的预后以及更长的生存时间,而且与患者不同的临床状态有密切关系; 与T细胞、Treg细胞、巨噬细胞、NK细胞等免疫细胞的浸润有一定相关性,说明FUT8可能是一个评价预后和免疫细胞浸润水平的标志物。
-
表 1 26个线粒体自噬相关基因列表
基因名称 基因中文译名 基因名称 基因中文译名 ATG5 自噬相关5同源物 SQSTM1 死骨片1 FUNDC1 含FUN14域1 UBB 泛素B CSNK2A2 酪蛋白激2 α2原肽 MFN1 丝裂融蛋白1 TOMM22 线粒体外膜转位酶22 TOMM20 线粒体外膜转位酶20 CSNK2A1 酪蛋白激酶2α1肽 TOMM5 线粒体外膜转位酶5 MAP1LC3A 微管关联蛋白1轻链3α ULK1 unc-51样激酶1 MFN2 丝裂融蛋白2 TOMM7 线粒体外膜转位酶7 TOMM40 线粒体外膜转位酶40 SRC 肉瘤病毒癌基因同源物 MAP1LC3B 微管关联蛋白1轻链3β CSNK2B 酪蛋白激酶2β肽 RPS27A 核糖体蛋白S27a VDAC1 电压依赖阴离子通道1 ATG12 自噬相关12同源物 TOMM6 线粒体外膜转位酶6 UBC 泛素C UBA52 泛素A-52 PINK1 PTEN引导预测激酶1 PGAM5 磷酸甘油酸变旋酶家族成员5 表 2 线粒体自噬预后相关基因的差异表达情况分析
基因名称 中文译名 log2FC P P.adj ATG12 自噬相关12同源物 -0.025 0.510 0.626 CSNK2A2 酪蛋白激酶2 α1肽 0.080 0.024 0.054 CSNK2B 酪蛋白激酶2 β肽 0.115 0.055 0.107 FUNDC1 含FUN14域1 0.011 0.867 0.915 MAP1LC3A 微管关联蛋白1轻链3α -0.100 0.273 0.391 MAP1LC3B 微管关联蛋白1轻链3β -0.151 0.001 0.005 PGAM5 磷酸甘油酸变旋酶家族成员5 0.430 < 0.001 < 0.001 PINK1 PTEN引导预测激酶1 -0.632 < 0.001 < 0.001 SQSTM1 死骨片1 0.115 0.073 0.136 TOMM20 线粒体外膜转位酶20 0.265 < 0.001 < 0.001 TOMM22 线粒体外膜转位酶22 -0.023 0.635 0.735 TOMM40 线粒体外膜转位酶40 0.328 < 0.001 < 0.001 TOMM5 线粒体外膜转位酶5 0.195 < 0.001 0.001 UBA52 泛素A-52 -0.039 0.435 0.554 UBC 泛素C -0.046 0.330 0.450 表 3 线粒体自噬预后相关基因的GO富集分析结果
类别 基因编码 描述 基因比值 背景比值 P P.adj q 生物学过程 GO: 0016236 大分子自噬 13/15 295/18 670 < 0.001 < 0.001 < 0.001 生物学过程 GO: 0000422 线粒体自噬 7/15 76/18 670 < 0.001 < 0.001 < 0.001 生物学过程 GO: 0006605 蛋白质定位 8/15 432/18 670 < 0.001 < 0.001 < 0.001 细胞成分 GO: 0005741 线粒体外膜 9/15 178/19 717 < 0.001 < 0.001 < 0.001 细胞成分 GO: 0098799 线粒体外膜蛋白复合体 4/15 16/19 717 < 0.001 < 0.001 < 0.001 细胞成分 GO: 0044455 线粒体膜部分 6/15 229/19 717 < 0.001 < 0.001 < 0.001 分子功能 GO: 0031625 泛素蛋白连接酶结合 6/14 290/17 697 < 0.001 < 0.001 < 0.001 分子功能 GO: 0008320 蛋白跨膜转运活性 3/14 20/17 697 < 0.001 < 0.001 < 0.001 分子功能 GO: 0004674 蛋白质丝氨酸/苏氨酸激酶活性 4/14 439/17 697 < 0.001 0.002 0.001 GO: 基因本体论; 基因比值: 目标基因富集到目的通路的基因个数占目标基因总数(包含基因集总基因)的比值; 背景比值: 参与该代谢通路的基因数与整个基因组中相关基因数的比值; q: 检测阳性个体中,假阳性个体所占比值。 表 4 线粒体自噬预后相关基因的KEGG通路富集分析结果
编码 描述 基因比值 背景比值 P P.adj q hsa04137 线粒体自噬-动物 8/12 68/8 076 < 0.001 < 0.001 < 0.001 hsa05022 神经退行性疾病-多种疾病的通路 7/12 475/8 076 < 0.001 < 0.001 < 0.001 hsa05131 志贺菌病 4/12 246/8 076 < 0.001 0.003 0.001 hsa04216 铁凋亡 2/12 41/8 076 0.002 0.011 0.005 hsa04520 黏着连接 2/12 71/8 076 0.005 0.024 0.010 hsa05012 帕金森病 3/12 249/8 076 0.005 0.024 0.010 hsa05014 肌萎缩侧索硬化症 3/12 364/8 076 0.015 0.038 0.016 hsa04140 自噬-动物 2/12 137/8 076 0.017 0.038 0.016 KEGG: 京都基因与基因组百科全书。 -
[1] SHAFABAKHSH R, YOUSEFI B, ASEMI Z, et al. Chitosan: a compound for drug delivery system in gastric cancer-a review[J]. Carbohydr Polym, 2020, 242: 116403. doi: 10.1016/j.carbpol.2020.116403
[2] 钱慧琴. 基于网络药理学探讨肿节风治疗胃癌的作用机制[J]. 沈阳药科大学学报, 2022, 39(8): 979-987. https://www.cnki.com.cn/Article/CJFDTOTAL-SYYD202208012.htm [3] AVENTAGGIATO M, VERNUCCI E, BARRECA F, et al. Sirtuins' control of autophagy and mitophagy in cancer[J]. Pharmacol Ther, 2021, 221: 107748. doi: 10.1016/j.pharmthera.2020.107748
[4] 李祎龙, 雷凯, 张小昭, 等. 1, 25-二羟维生素D3对人结肠癌细胞凋亡和自噬的影响及机制研究[J]. 实用临床医药杂志, 2021, 25(13): 5-9. doi: 10.7619/jcmp.20211388 [5] FERRO F, SERVAIS S, BESSON P, et al. Autophagy and mitophagy in cancer metabolic remodelling[J]. Semin Cell Dev Biol, 2020, 98: 129-138. doi: 10.1016/j.semcdb.2019.05.029
[6] 张标. TP53突变在肝细胞癌中的意义[D]. 大连: 大连医科大学, 2022. [7] YUAN Q H, DENG D W, PAN C, et al. Integration of transcriptomics, proteomics, and metabolomics data to reveal HER2-associated metabolic heterogeneity in gastric cancer with response to immunotherapy and neoadjuvant chemotherapy[J]. Front Immunol, 2022, 13: 951137. doi: 10.3389/fimmu.2022.951137
[8] GOLDMAN M J, CRAFT B, HASTIE M, et al. Visualizing and interpreting cancer genomics data via the Xena platform[J]. Nat Biotechnol, 2020, 38(6): 675-678. doi: 10.1038/s41587-020-0546-8
[9] 张良舜, 叶桦. 基于GEO胃癌芯片数据的生物信息学分析[J]. 现代实用医学, 2022, 34(11): 1409-1411, F0002. doi: 10.3969/j.issn.1671-0800.2022.11.004 [10] 唐焘, 武敏, 王君, 等. C型尼曼-匹克蛋白2在胃癌组织中的表达及其功能初步研究[J]. 第三军医大学学报, 2021, 43(21): 2381-2388. https://www.cnki.com.cn/Article/CJFDTOTAL-DSDX202121017.htm [11] 蒿花, 田国祥, 耿辉, 等. 人类基因综合分析数据库GeneCards的应用介绍[J]. 中国循证心血管医学杂志, 2021, 13(8): 902-906. doi: 10.3969/j.issn.1674-4055.2021.08.03 [12] 葛继芸, 姚晨, 张菁, 等. CALB2介导的肿瘤微环境免疫细胞浸润在肝癌复发中的作用机制分析[J]. 海军军医大学学报, 2022, 43(7): 744-751. https://www.cnki.com.cn/Article/CJFDTOTAL-DEJD202207004.htm [13] 桑珍珍, 杨栋梁, 饶欣, 等. 基于加权基因共表达网络分析探讨新型冠状病毒感染相关脓毒症潜在的关键基因[J]. 中国急救医学, 2023, 43(5): 376-382. https://www.cnki.com.cn/Article/CJFDTOTAL-ZJJY202305007.htm [14] 何融泉. 大数据先验模式构建膀胱癌非编码RNA全景图谱及SCARNA12的分子机制研究[D]. 南宁: 广西医科大学, 2019. [15] 吴茜, 宋兴勃, 钟慧钰, 等. 胃癌关键基因和通路的生物信息学和功能分析[J]. 肿瘤预防与治疗, 2020, 33(2): 131-139. https://www.cnki.com.cn/Article/CJFDTOTAL-SCZF202002009.htm [16] 姚志强. 基于生物信息学鉴定CTSV作为膀胱癌诊断和预后的标志物及预后nomogram的构建[D]. 兰州: 兰州大学, 2021. [17] 罗倩, 詹雪冰, 董芳媛, 等. 基于数据库分析CDCA基因家族在胃癌中的表达及预后意义[J]. 包头医学院学报, 2021, 37(11): 45-50. https://www.cnki.com.cn/Article/CJFDTOTAL-BTYX202111011.htm [18] 张颖, 邱汉波, 侯恩存, 等. 基于网络药理学-分子对接研究益气逐淤汤针对胃癌的作用机制[J]. 世界中医药, 2021, 16(23): 3484-3490. https://www.cnki.com.cn/Article/CJFDTOTAL-SJZA202123014.htm [19] NIE Y L, LIANG X J, LIU S H, et al. WASF3 knockdown sensitizes gastric cancer cells to oxaliplatin by inhibiting ATG12-mediated autophagy[J]. Am J Med Sci, 2020, 359(5): 287-295.
[20] MENG L, HU P B, XU A M. PGAM5 promotes tumorigenesis of gastric cancer cells through PI3K/AKT pathway[J]. Pathol Res Pract, 2023, 244: 154405.
[21] 张浩, 张悦, 赵文武, 等. PINK1/Parkin介导的线粒体自噬及其在肝脏疾病发生发展中的作用机制[J]. 临床肝胆病杂志, 2020, 36(7): 1663-1665. https://www.cnki.com.cn/Article/CJFDTOTAL-LCGD202007057.htm [22] 肖依依. 二甲双胍通过激活AMPKα诱导PINK1/Parkin介导的线粒体自噬降低胃癌细胞顺铂化疗敏感性的研究[D]. 南昌: 南昌大学, 2022. [23] SALAZAR C, RUIZ-HINCAPIE P, RUIZ L. The interplay among PINK1/PARKIN/dj-1 network during mitochondrial quality control in cancer biology: protein interaction analysis[J]. Cells, 2018, 7(10): 154.
[24] MIYAZAKI N, SHIRATORI R, OSHIMA T, et al. PINK1-dependent and Parkin-independent mitophagy is involved in reprogramming of glycometabolism in pancreatic cancer cells[J]. Biochem Biophys Res Commun, 2022, 625: 167-173.
[25] 李娜, 王培红, 李俊杰, 等. MIF通过调节急性幽门螺杆菌感染介导的炎症反应、自噬和凋亡促进胃癌细胞增殖及细胞周期进展[J]. 中国免疫学杂志, 2023, 39(2): 336-342. https://www.cnki.com.cn/Article/CJFDTOTAL-ZMXZ202302019.htm





 下载:
下载:






 苏公网安备 32100302010246号
苏公网安备 32100302010246号
